La pandémie liée à SARS-CoV-2 pose de façon aiguë la question du design de molécules capables de limiter l’action d’un virus sur nos cellules – un mécanisme qui implique des molécules de très grande taille difficiles à modéliser, des protéines, qui sont de plus… en mouvement permanent.
Des systèmes d’intelligence artificielle, à commencer par AlphaFold2 de Google, prédisent désormais la configuration de ces protéines de façon impressionnante, ce qui révolutionne la recherche dans le domaine. Comment marchent ces méthodes ? Quelles sont leurs limites actuelles ?
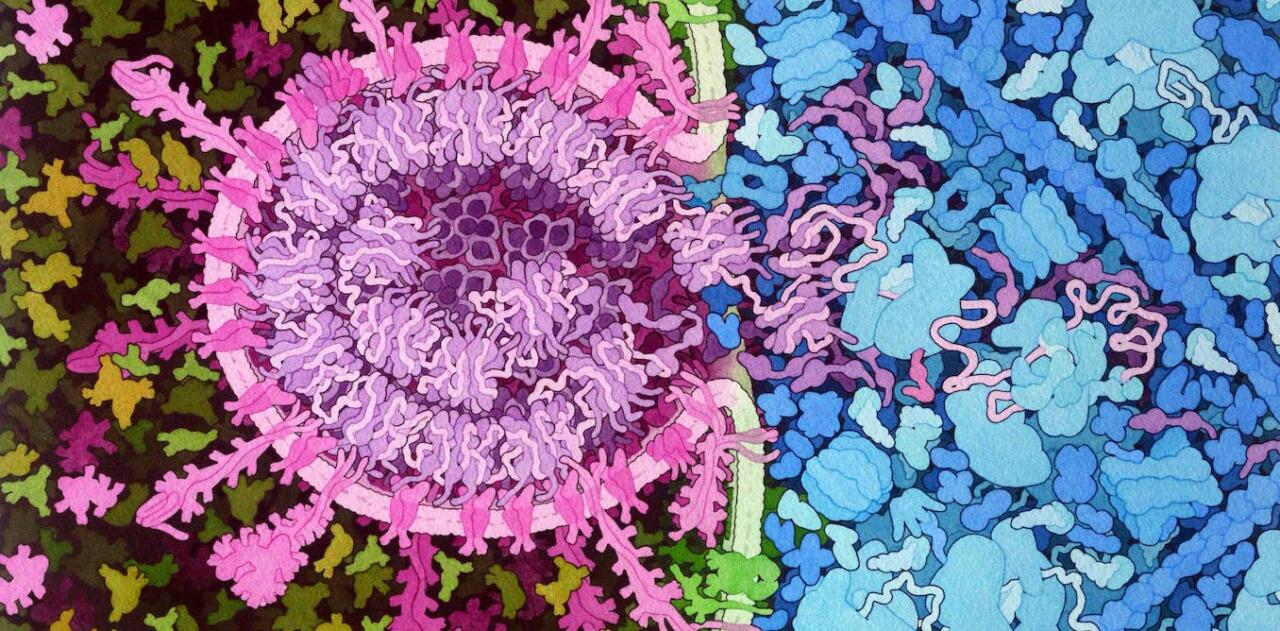
Entrée de SARS-CoV-2 : une histoire de monte-en-l’air
L’infection de l’une de nos cellules par le SARS-CoV-2, le virus responsable du Covid-19, commence par une sorte d’effraction : le virus, une enveloppe hérissée de protéines à l’intérieur de laquelle se trouve son matériel génétique, se comporte comme un voleur entrant dans un appartement au premier étage d’un immeuble. Avec un grappin (le « domaine de fixation au récepteur » ou RBD se trouvant sur la fameuse protéine « spike »), il s’accroche à la rambarde (la non moins fameuse protéine « ACE2 »). Puis, à l’aide d’un marteau (le domaine de fusion, une autre région du spike), il brise la vitre (la membrane de la cellule) et injecte son matériel génétique.
Ce mécanisme est dynamique, c’est-à-dire que les molécules changent de conformation (de forme) pendant l’effraction. D’une part, le virus ne « dégaine » son grappin qu’au dernier instant ; d’autre part, le « bris de vitre » utilise une sorte de perche télescopique dont l’assemblage est complexe.
Fusion de la membrane de la cellule et du virus ; source : ClarafiSciViz.
Ces deux protagonistes (spike et ACE2) sont des protéines. Les interactions entre protéines sont à la base de l’immense majorité des fonctions biologiques, et la compréhension de ces interactions nécessite dans un premier temps la connaissance de la forme géométrique des partenaires – on parle souvent de « clef » et de « serrure » pour visualiser le fait que la géométrie des protéines doit être adéquate pour qu’elles interagissent.
Ces conformations moléculaires sont étudiées expérimentalement depuis les années 1950-60 et entreposées dans une base de données internationale, la Protein Data Bank.
[Près de 80 000 lecteurs font confiance à la newsletter de The Conversation pour mieux comprendre les grands enjeux du monde. Abonnez-vous aujourd’hui]
Dans le cas du SARS-CoV-2, on a beaucoup présenté la protéine Spike comme une telle clef, qui s’emboîterait dans la « serrure » ACE2. Mais le mécanisme clef-serrure est une vision un peu simpliste, et comme on l’a vu les protéines sont dotées d’une certaine flexibilité (elles se déforment), ce qui leur…
La suite est à lire sur: theconversation.com
Auteur: Frédéric Cazals, Directeur de recherche; Equipe Algorithms-Biology-Structure, Inria